Elasticsearch 向量检索相关知识点学习
参考资料
基本知识
向量数据库主要用于存储和查询高维向量,其核心功能是支持高效的向量相似度搜索。其中的知识点如下:
如何衡量 2 个向量相似?参考欧氏距离和余弦相似度
- l2_norm,欧式距离,公式是
(x1-y1)^2+...+(xk-yk)^2的平方根 - cosine,余弦相似度,公式是
x1*y1+...+xk*yk除以||A||*||B|| - dot_product,点积,公式是
x1*y1+...+xk*yk
向量检索方式,假设向量维度为k,数量为n:
- Brute-force,暴力检索,时间复杂度是O(n)
- k-dimension tree,基于特殊的二叉树,时间复杂度是O(log(n))
- IVF(Inverted File System)
- PQ(Product Quantizer)
- IVF-PQ(Inverted File System - Product Quantizer)
- LSH(Locality Sensitive Hashing)
- HNSW(Hierarchical Navigable Small Worlds)
以上除了暴力检索,其他都属于ANN(Approximate Nearest Neighbor)的相似性检索算法,大体可以分为三大类:
- 基于树的方法,低维度的数据性能比较高效;高维度的数据性能较差
- 哈希方法,对于小数据集和中等数据集效果不错
- 矢量量化,对于大规模数据集,矢量量化是个很好的选择
Brute-force
检索时,依次对每个数据进行相似度计算,返回最接近的值
k-dimension tree
存储过程:在第一维取中位值为根节点,分成左右两边数据,左边数据小于根节点,右边数据大于根节点。进入第二维,左右两边取第二维的中位值作为根节点,分成左右两边数据,此时第一维根节点分别指向左边根节点和右边根节点。依次递归,只到完成所有维度。代码实现如下:
class KDTreeNode:
def __init__(self, point, split_dim=None, left=None, right=None):
self.point = point
self.split_dim = split_dim
self.left = left
self.right = right
def build_kdtree(points, depth=0):
n = len(points)
if n == 0:
return None
k = len(points[0]) # Dimensionality of the points
# Select the splitting dimension
split_dim = depth % k
# Sort points along the selected dimension
points.sort(key=lambda point: point[split_dim])
# Choose the median point
median = n // 2
# Create the node and recursively build the left and right subtrees
return KDTreeNode(
point=points[median],
split_dim=split_dim,
left=build_kdtree(points[:median], depth + 1),
right=build_kdtree(points[median + 1:], depth + 1)
)
# Example usage:
points = [(2, 3), (5, 4), (9, 6), (4, 7), (8, 1), (7, 2)]
tree = build_kdtree(points)IVF
先通过聚类算法把数据分成m个子空间,检索的时候看查询的vector和那个子空间最相近,然后只在这个子空间内进行检索。代码实现如下:
import numpy as np
from sklearn.cluster import KMeans
def cosine_similarity(x, y):
num = x.dot(y.T)
norm = np.linalg.norm(x) * np.linalg.norm(y)
return num / norm
class VectorSpaceSearch:
def __init__(self, n_clusters):
self.n_clusters = n_clusters
self.kmeans = KMeans(n_clusters=n_clusters)
self.inverted_index = {}
def fit(self, X):
"""Fit the model to the data X and build the inverted index."""
labels = self.kmeans.fit_predict(X)
for i, label in enumerate(labels):
if label not in self.inverted_index:
self.inverted_index[label] = []
self.inverted_index[label].append(X[i])
def search(self, query, similarity_func):
"""Search for the most similar vector to the query."""
label = self.kmeans.predict(query.reshape(1, -1))
cluster = self.inverted_index[label[0]]
similarities = [similarity_func(query, x) for x in cluster]
return cluster[np.argmax(similarities)]
data = np.array([(2, 3), (5, 4), (9, 6), (4, 7), (8, 1), (7, 2)])
vss = VectorSpaceSearch(3)
vss.fit(data)
query = np.array([(2, 3.1)])
result = vss.search(query, cosine_similarity)
print(result)PQ
假设向量个数有N个,维度是128,维度切成M份,这样每份的维度就是128/M。然后每份又通过聚类算法分成256簇。每来一个查询向量,也先切成M份,再分别与这些簇的中心点计算距离,例如第一条距离等于l1+…+lM。得到256条结果,取距离最近,得到M个簇,计算查询向量与这M个簇的簇内向量距离,取topN。
IVF-PQ
参考PQ和IVF介绍和IVF-PQ算法与faiss源码分析。假设簇心个数为1024,那么每来一个查询向量,首先计算其与1024个粗聚类簇心的距离,然后选择距离最近的top N个簇,只计算查询向量与这几个簇底下的向量的距离,计算距离的方法就是前面提到的PQ。
LSH
假设有数据集合中有6个点:A=(1,1) B=(2,1) C=(1,2) D=(2,2) E=(4,2) F=(4,3),01编码后是:
v(A)=10001000
v(B)=11001000
v(C)=10001100
v(D)=11001100
v(E)=11111100
v(F)=11111110
这里有2个参数:
L=3,表示hash表的个数
K=2,表示二进制的位数,它指示了每个hash表的簇数是K^2
假设3个hash表对应的3个hash函数如下:
g1分别取第2位,第4位
g2分别取第1位,第6位
g3分别取第3位,第8位
以元素A为例,按照规则g1(A)=00,g2(A)=10,g3(A)=00,其它依次类推,最后得到:
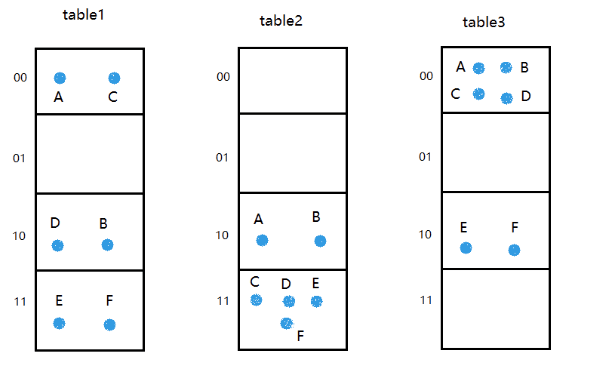
设查询向量q=(4,4)=(11111111),可以计算出g1(q)=11,g2(q)=11,g3(q)=11,然后从3个hash表对应的簇中取出所有元素,即C,D,E,F。
HNSW
思路与常见的跳表算法相似,如下图要搜索跳表,从最高层开始,沿着具有最长“跳过”的边向右移动。如果发现当前节点的值大于要搜索的值-我们知道已经超过了目标,因此我们会在下一级中向前一个节点。
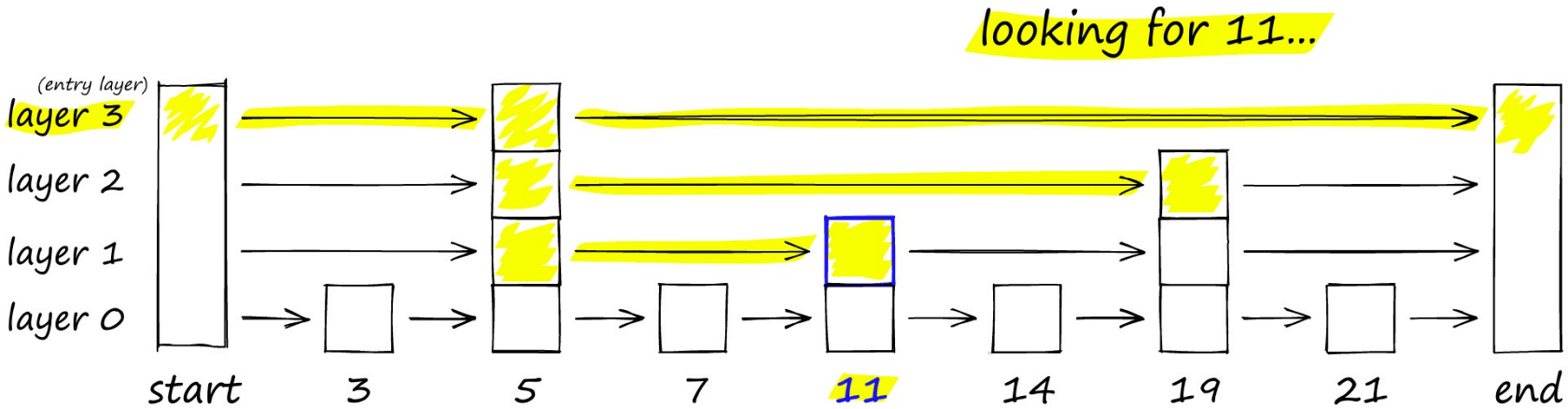
HNSW 算法是一种经典的空间换时间的算法,它的搜索质量和搜索速度都比较高,但是它的内存开销也比较大,因为不仅需要将所有的向量都存储在内存中。还需要维护一个图的结构,也同样需要存储。所以这类算法需要根据实际的场景来选择。
本作品采用《CC 协议》,转载必须注明作者和本文链接



 关于 LearnKu
关于 LearnKu



